Benjamin R. Evans ,
Panayiota Kotsakiozi ,
André Luis Costa-da-Silva ,
Rafaella Sayuri Ioshino ,
Luiza Garziera ,
Michele C. Pedrosa ,
Aldo Malavasi ,
Jair F. Virginio ,
Margareth L. Capurro e
Jeffrey R. Powell
Relatórios Científicos volume 9 , Artigo número: 13047 ( 2019 ) Citar este artigo
Tradução Ricardo Camillo
Resumo
Em uma tentativa de controlar as doenças transmitidas por mosquitos, febre amarela, dengue, chikungunya e febres do Zika, uma cepa do Aedes aegypti modificado transgenicamente mosquitos contendo um gene letal dominante foram desenvolvidos por uma empresa comercial, a Oxitec Ltd. Se a letalidade for completa, a liberação dessa cepa deve apenas reduzir o tamanho da população e não afetar a genética das populações-alvo. Aproximadamente 450 mil machos dessa cepa foram liberados a cada semana durante 27 meses em Jacobina, Bahia, Brasil. Nós genotipamos a cepa de liberação e a população alvo de Jacobina antes do início das liberações para> 21.000 polimorfismos de nucleotídeo único (SNPs). A amostragem genética da população-alvo seis, 12 e 27-30 meses após o início das liberações fornece evidências claras de que partes do genoma da cepa transgênica foram incorporadas à população-alvo. Evidentemente, raros descendentes de híbridos viáveis entre a cepa de liberação e a população Jacobina são suficientemente robustos para serem capazes de se reproduzir na natureza. A cepa de liberação foi desenvolvida usando uma cepa originária de Cuba, depois cruzada com uma população mexicana. Assim, JacobinaAe. aegypti agora é uma mistura de três populações. Não está claro como isso pode afetar a transmissão de doenças ou afetar outros esforços para controlar esses vetores perigosos. Esses resultados destacam a importância de haver um programa de monitoramento genético durante essas liberações para detectar resultados inesperados.
Introdução
As doenças transmitidas por mosquitos afetam enormemente a saúde humana e as economias, especialmente nos países do Terceiro Mundo. Vacinas e medicamentos eficazes estão disponíveis para apenas alguns, então o principal meio de controlar essas doenças é controlar os mosquitos que as transmitem. Como os métodos tradicionais de controle, como os inseticidas, tornaram-se menos eficazes e aceitáveis, métodos alternativos têm sido buscados 1 . Métodos baseados em manipulações genéticas estão entre os mais atraentes e ativamente perseguidos 2 . Um desses programas de base genética envolveu a liberação de uma cepa de Aedes aegypti (OX513A) que foi transgenicamente modificada para ser homozigótica para um letal dominante condicional 3 , 4. Esta cepa também carrega um gene de proteína fluorescente que permite a detecção da descendência F 1 de tipo selvagem OX513A X. A liberação dessa cepa em grande número tem sido eficaz na redução das populações de Ae. aegypti em até 85% 5 . Os maiores lançamentos até hoje foram realizados na cidade de Jacobina, na Bahia, Brasil 6 . Monitoramos a Jacobina Ae. aegyptipopulação para determinar se as liberações afetaram a genética da população natural por meio da transferência de genes, introgressing. Se a letalidade for completa, essas liberações devem resultar apenas na redução da população e não afetar a genética da população-alvo. No entanto, sabe-se que, em condições de laboratório, 3-4% dos descendentes de cruzamentos de OX513A com o tipo selvagem sobrevivem até a idade adulta, embora sejam fracos e não se sabe se são férteis 4 .
Materiais e métodos
Locais de soltura e criação
Jacobina, no estado da Bahia, Brasil, é uma cidade de tamanho moderado de ~ 75.000 habitantes localizada nas coordenadas 11 ° 10′51 ″ S, 40 ° 31′04 ″ W (Fig. 1 ). Jacobina é cercada por vários quilômetros em todas as direções pela caatinga, um bioma ecológico seco no qual Ae. aegypti não pode se reproduzir, tornando Jacobina uma ilha para esse mosquito.
figura 1
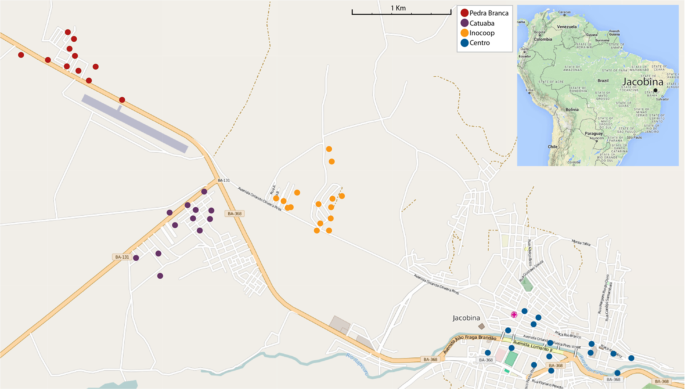
Mapa de Jacobina. As ovitrampas onde as amostras foram coletadas são indicadas com pontos coloridos, codificados por bairro. Os lançamentos foram feitos nos bairros de Pedra Branca, Catuaba e Inocoop, mas nunca na área Centro. Contribuidores do © OpenStreetMap.
Imagem em tamanho grande
O criadouro da linhagem de liberação está localizado na Biofabrica Moscamed Brasil em Juazeiro, cerca de 200 quilômetros ao norte de Jacobina. A criação em massa e a sexagem são descritas em Harris et al . 7 . Semanalmente, pupas machos eram transportadas para Jacobina e mantidas em uma instalação local por uma semana para permitir a eclosão antes da liberação; aproximadamente 450 mil machos OX513A foram lançados a cada semana a partir de junho de 2013 e continuaram até setembro de 2015 6 . Os lançamentos foram feitos nos bairros Pedra Branca, Catuaba e Inocoop, mas nunca no Centro. As armadilhas de oviposição foram amostradas semanalmente nas localidades indicadas na Fig. 1 . Os ovos foram chocados e as frequências de larvas fluorescentes e selvagens registradas; ver Garzeira et al. 6 para obter detalhes de proporção fluorescente e tipo selvagem em cada ponto de tempo. As larvas de quarto instar de cada tipo foram colocadas em ~ 80% de etanol e levadas para a Universidade de Yale para genotipagem. Mais dados sobre o efeito de liberações em Jacobina podem ser encontrados em Graziera et al . 6 .
Análises genéticas
Usamos um chip SNP Affymetrix desenvolvido para genotipagem 8 . Aproximadamente 200 ng de DNA genômico de mosquitos individuais foram colocados em 95 poços de uma placa de 96 poços, com um controle de água destilada. As placas foram enviadas para o Functional Genomics Core na University of North Carolina, Chapel Hill, para hibridização e produção de arquivos de dados enviados para a Yale University. Usamos o pacote R SNPolisher v1.4 (Afffymetrix, Santa Clara, CA) para gerar e processar chamadas de genótipo. Enquanto o chip SNP contém sondas para cerca de 27.000 SNPs bialélicos bem validados, passando em testes de herança Mendeliana e genotipagem> 98% de todas as amostras 8 , 21.770 foram polimórficos em nossas amostras de Jacobina e genotipados em> 98% de todos os indivíduos.
Nós genotipamos amostras retiradas do Centro e uma amostra combinada de Catuaba / Pedra Branca antes do início das liberações. Então, enquanto os lançamentos continuavam, amostramos todos os bairros seis, 12 e 27–30 meses após o início dos lançamentos. A última amostra em 27-30 meses foi uma amostra combinada de três meses, incluída depois que as liberações cessaram aos 27 meses. Os tamanhos das amostras estão na Tabela 1 . Exceto para a amostra final combinada de 27-30 meses, cada amostra analisada após o início das liberações era de armadilhas de ovos expostas por uma única semana e amostras de larvas de pelo menos cinco armadilhas em cada bairro. A posição das armadilhas permaneceu a mesma ao longo do estudo.
Tabela 1 Resultados da análise “INTROGRESS” realizada com o pacote R 10 .
Mesa em tamanho grande
Para confirmar que nossas análises genéticas foram precisas na detecção de híbridos, também genotipamos 57 larvas fluorescentes coletadas seis meses nas liberações que representam a prole F 1 entre a cepa de liberação e a população natural.
Análises
Realizamos três tipos de análises. Primeiro, para confirmar que nosso painel de SNPs poderia discriminar entre a cepa de liberação OX513A e a população natural antes da liberação, realizamos uma Análise de Componentes Principais (PCA) usando o pacote R em LEA 9 . Em segundo lugar, o pacote R “introgresso” 10 foi implementado designando OX513A e Jacobina antes do lançamento (bairros Centro, Catuaba e Pedra Branca combinados) como as duas populações parentais. Terceiro, realizamos uma análise ADMIXTURE conforme descrito em 11 e mostrado na Fig. 2C . Para esta análise, filtramos para excluir SNPs fortemente vinculados usando a opção –indep do PLINK 12resultando em um painel de 14.252 SNPs. Em seguida, uma análise ANOVA seguida por um teste post-hoc de TukeyHSD foi usada para testar as diferenças estatísticas (nível de confiança de 0,95) nos valores médios de Q entre as populações e, mais importante, entre as populações pré e pós-liberação.
Figura 2
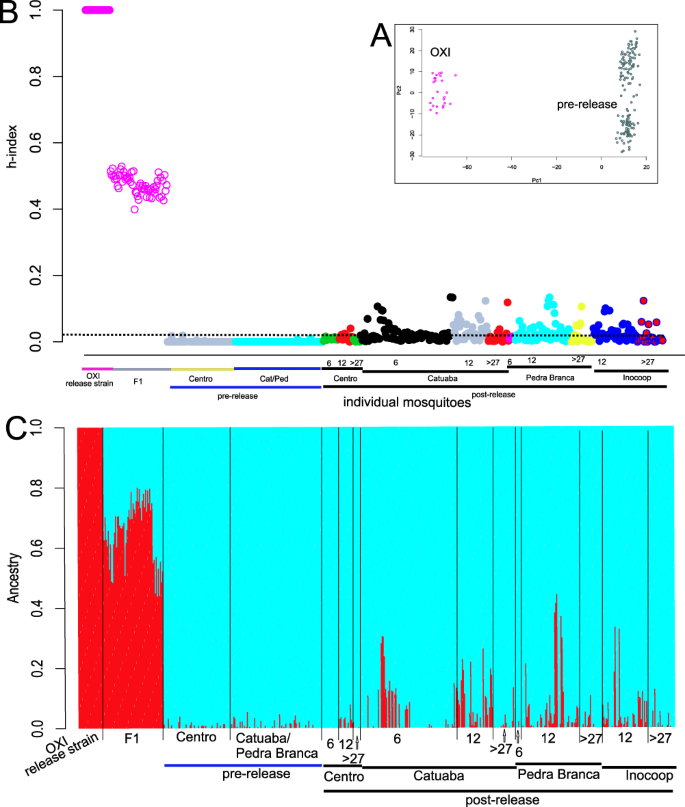
( A ) Análise de Componentes Principais (PCA) na cepa de liberação OX513A e três bairros Jacobina (Centro e Catuaba / Pedra Branca) antes do início das liberações. ( B ) Índice híbrido (índice h) conforme executado em INTROGRESS 10 . Um índice de 1,0 indica os indivíduos OX513A “puros”, 0,0 indica os indivíduos Jacobina “puros” em pré-lançamento. Os indivíduos são organizados por bairro indicado na parte inferior da figura e, a seguir, por data de coleta: pré-lançamento, 6, 12 ou 27–30 meses após o lançamento. Os híbridos F1 verificados por fluorescência são agrupados e rotulados como F1. A linha tracejada horizontal representa o ponto de corte (índice h = 0,02) do pré-lançamento máximo observado. ( C ) ADMIXTURE 11análise de todos os genótipos individuais. A proporção de cada cor para cada indivíduo representa a proporção da ancestralidade desse indivíduo atribuível ao aglomerado vermelho (OX513A) ou azul (pré-lançamento Jacobina).
Imagem em tamanho grande
Infecções de vírus
A cepa testada do vírus da dengue sorotipo 2 (DENV-2) foi isolada durante uma epidemia no Brasil em 2010 de um paciente em Santos, Brasil. A cepa, designada ACS46 13 , foi descrita em Cugola et al . 14 e gentilmente cedido pelo Instituto Evandro Chagas de Belém-Pará.
Os procedimentos de infecção do mosquito são descritos em detalhes em Cost-da-Silva et al . 15 . Resumidamente, fêmeas pré-acasaladas de cinco a sete dias de idade foram alimentadas com sangue artificialmente usando o alimentador Glytube (22). DENV-2 da nona subcultura (T9) ou ZIKV BR da quarta subcultura (T4) foram misturados com concentrado de eritrócito humano e soro sanguíneo inativado para alimentar as fêmeas. As concentrações finais de DENV-2 e ZIKV BR na solução de alimentação foram 1,7 × 10 10 cópias do genoma / ml e 2,2 × 10 6 unidades formadoras de placa (ufp) / ml, respectivamente.
Ensaios de vírus
Fêmeas ingurgitadas das cepas ROCK, OX513A e Jacobina foram separadas dos mosquitos não ingurgitados e mantidas em sacarose a 10%. Quatorze dias após a refeição de sangue (14 PBM), as fêmeas foram anestesiadas com CO 2 e mantidas em gelo. Os corpos individuais dos mosquitos foram separados das cabeças e congelados separadamente imediatamente em gelo seco e armazenados a -80 ° C. O RNA total foi extraído usando QIAamp Viral RNA Mini Kit (Qiagen). As cópias genômicas de DENV-2 ou ZIKV BR foram medidas usando o método de qRT-PCR de uma etapa, conforme descrito em (22). Para gerar a curva padrão DENV-2, um fragmento de 119 pb da cepa ACS46 foi amplificado com os iniciadores D1-TS2 15e foi clonado no vetor pCR2.1 (Invitrogen). Este plasmídeo foi usado para estimar o número de cópias de DENV para cada amostra. As condições do termociclador para amplificação do DENV-2 foram 48 ° C por 30 min e 95 ° C por 10 min; 45 ciclos de 95 ° C por 30 seg, 55 ° C por 30 seg e 60 ° C por 30 seg, e uma curva de fusão de 95 ° C por 1 min, 60 ° C por 30 seg e 95 ° C por 1 min , com temperatura subindo de 60 ° C a 95 ° C a 0,02 ° C / s.
Análises estatísticas foram realizadas para avaliar diferenças significativas nos níveis virais (teste de Kruskal-Wallis seguido pelo Teste de Comparação Múltipla de Dunn) ou taxas de infecção de cabeças ou corpos (teste exato de Fisher) entre as três cepas de mosquitos. O programa e os procedimentos para realização das análises foram descritos anteriormente 15 .
Resultados
A Figura 2A mostra que nossos 21.770 SNPs distinguem claramente OX513A e a população natural de Jacobina. Na Fig. 2B, C fica claro que os três bairros antes das liberações, Pedra Branca, Catuaba e Centro, são geneticamente bastante homogêneos; ou seja, não há indicação de heterogeneidade genética em Ae. aegypti coleta amostras ao longo de aproximadamente 6 km de extensão da cidade (Fig. 1 ) antes do início das liberações. A Figura 2B, C também indica que podemos identificar a prole F 1 entre a cepa de liberação e a população natural em Jacobina.
Para detectar a introgressão, genotipamos um total de 347 Ae de tipo selvagem (não fluorescente) . aegypti em Jacobina amostrada 6 meses, 12 meses e 27-30 meses após o início das liberações. A Figura 2B, C indicam claramente os mosquitos individuais com genomas mistos nos bairros onde as liberações foram feitas. Mesmo no bairro onde não foram realizados lançamentos, Centro, algum grau de introgressão pode ser detectado, possivelmente devido à migração dos bairros de lançamento cerca de quatro quilômetros de distância (Fig. 1 ). Na Tabela 1, apresentamos dados numéricos incluindo tamanhos de amostra (entre parênteses) para cada amostra em cada localidade em cada momento. Usamos dois pontos de corte indicando indivíduos introgressados inequívocos: h = 0,02 o máximo observado antes das liberações (também a linha pontilhada na Fig. 2B) eh = 0,04 o máximo encontrado após lançamentos no Centro, onde não foram realizados lançamentos. No critério menos rigoroso, entre cerca de 20 e 60% dos mosquitos amostrados foram introgressados; para o critério mais rigoroso, cerca de 5 a 30% são introgressados. A introgressão máxima (valor h) possível é 25%, retrocruzamento de primeira geração. O máximo que observamos foi 0,13 indicativo de retrocruzamento de segunda geração; nossa primeira amostra em seis meses é tempo suficiente para produzir múltiplas gerações de retrocruzamento, dado um tempo de geração de cerca de um mês. Espera-se que a primeira progênie de retrocruzamento seja mais rara do que as gerações posteriores, então não é surpreendente que apenas uma única progênie de retrocruzamento de segunda geração tenha sido observada com a maioria retrocruzada avançada.
Os dados da Fig. 2 e da Tabela 1 são para todos os mosquitos amostrados. Também podamos os dados para controlar tamanhos de amostra desiguais e os resultados são semelhantes, com, de fato, mais indivíduos acima dos pontos de corte devido provavelmente a grupos parentais mais homogêneos (Dados estendidos, Tabela E1 ). A frequência de amostragem de indivíduos introgressados aumentou entre as amostras aos seis meses e aos 12 meses, mas diminui um pouco aos 27 meses (Tabela 1 e Tabela de Dados Ampliados E2 ).
É difícil realizar testes estatísticos no índice h (Fig. 2B ), mas os gráficos de ESTRUTURA com valores Q (Fig. 2C ) permitem o teste estatístico. ANOVA seguido por um teste de TukeyHSD confirmou diferenças significativas (p <0,05) nos valores médios de Q de pré-lançamento em Catuaba em seis e 12 meses, e em 12 meses em Inocoop e Pedra Branca (Dados estendidos, Fig. E1 ).
Os resultados de nossos testes de infectividade de uma cepa de cada um dos vírus dengue e Zika em fêmeas da cepa OX513A e da população natural Jacobina (antes das liberações) não indicam diferenças significativas (Fig. 3 ).
Figura 3
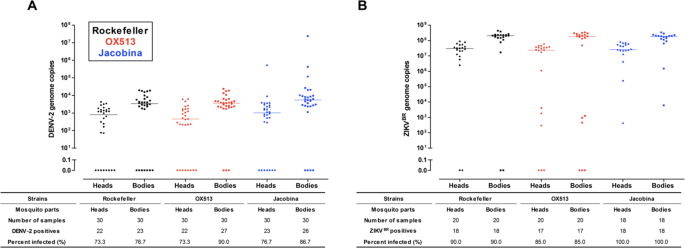
Níveis de cópias genômicas de DENV-2 (esquerda) e ZIKV (direita) detectados em cabeças e corpos sem cabeças de mosquitos Rockefeller, OX513 e Jacobina desafiados por infecção oral. Nenhuma das cepas difere pelos testes de Kruskal Wallis seguidos pelo pós-teste de Dunn (p> 0,05). Detalhes experimentais em materiais suplementares.
Imagem em tamanho grande
Discussão
Nossos dados mostram claramente que a liberação do OX513A levou a uma transferência significativa de seu genoma (introgressão) para a população natural de Jacobina de Ae. aegypti . O grau de introgressão não é trivial. Dependendo da amostra e do critério usado para definir introgressão inequívoca, cerca de 10% a 60% de todos os indivíduos têm algum genoma OX513A (Tabelas 1 e E1).
Uma aparente anomalia nos dados é a aparente diminuição na frequência de indivíduos introgressados entre a amostra de 12 meses e a amostra de 27-30 meses. No entanto, fica claro a partir dos dados de Garziera et al . 6 que a eficácia do programa de liberação começou a diminuir após cerca de 18 meses, ou seja, a população que havia sido bastante reprimida voltou a quase atingir os níveis anteriores à liberação. Especula-se que isso se deve à discriminação de acasalamento contra machos OX513A, um fenômeno conhecido por ocorrer em programas de liberação de machos estéreis 16 . Essa observação também implica que os indivíduos introgrados podem estar em uma desvantagem seletiva, causando sua aparente diminuição após o término da liberação, embora muito mais dados sejam necessários para confirmar isso.
Não se sabe o que impacta a introgressão de uma cepa transgênica de Ae. aegypti possui características importantes para o controle e transmissão de doenças. Testamos OX513A e Jacobina antes da liberação para taxas de infecção por uma cepa de cada um dos vírus da dengue e Zika e não encontramos diferenças significativas (Fig. 3 ). No entanto, isso é para apenas uma cepa de cada vírus em condições de laboratório; sob condições de campo para outros vírus, os efeitos podem ser diferentes. Além disso, a introgressão pode introduzir outros genes relevantes, como para a resistência a inseticidas. A cepa de liberação, OX513A, foi derivada de uma cepa de laboratório originalmente de Cuba, então cruzada com uma população mexicana 7. As três populações que formam a população tri-híbrida agora em Jacobina (Cuba / México / Brasil) são geneticamente muito distintas (dados estendidos Fig. E2 ), muito provavelmente resultando em uma população mais robusta do que a população de pré-liberação devido ao vigor do híbrido.
Esses resultados demonstram a importância de haver um programa de monitoramento genético durante a liberação de organismos transgênicos para detectar consequências inesperadas.
Histórico de mudanças
17 de setembro de 2019
Nota do Editor: os leitores são alertados de que as conclusões deste artigo estão sujeitas a críticas que estão sendo consideradas pelos editores. Uma resposta editorial adicional seguirá a resolução dessas questões.
14 de maio de 2020
Nota do Editor: uma Expressão de Preocupação editorial sobre este artigo foi agora publicada https://www.nature.com/articles/s41598-020-62398-w
Referências
1
Ferguson, NM Desafios e oportunidades no controle de infecções transmitidas por mosquitos. Nature 559 , 490 (2018).
PUBLICIDADES CAS Artigo Google Scholar
2
McGraw, EA & O'Neill, SL Além dos inseticidas: um novo pensamento sobre um problema antigo. Nature Rev Microbiol 11 , 181–193 (2013).
CAS Artigo Google Scholar
3
Thomas, DD, Donnell, CA, Wood, JJ & Alphey, LS Controle de população de insetos usando um sistema genético dominante, repressível e letal. Science 287 , 2474–2476 (2000).
PUBLICIDADES CAS Artigo Google Scholar
4
Phuc, HK et al . Sistemas genéticos letais dominantes de ação tardia e controle de mosquitos. BMC Biol 5 , 11 (2007).
Artigo Google Scholar
5
Carvalho, DO et al . Supressão de uma população de campo de Aedes aegypti no Brasil pela liberação sustentada de mosquitos machos transgênicos. PLoS Negl Trop Dis 9 (7), e0003864 (2015).
Artigo Google Scholar
6
Garziera, L. et al . Efeito da interrupção do alagamento remanescente de mosquitões transgênicos sobre a população silvestre de Aedes aegypi : dois estudos de caso no Brasil. Entomol. Eperimento. Appl. 164 , 327-339 (2017).
Artigo Google Scholar
7
Harris, A. et al . Supressão bem-sucedida de uma população de mosquitos do campo por meio da liberação sustentada de mosquitões machos manipulados. Nature Biotch. 30 , 828–830 (2002).
Artigo Google Scholar
8
Evans, BR et al . Um chip SNP multifuncional de alta capacidade para o mosquito da dengue e da febre amarela. Aedes aegypti. G3 (5), 711–718 (2015).
Google Scholar
9
Frichot, E. & Francois, O. LEA: um pacote R para os estudos da Paisagem e da Associação Ecológica. Methods Ecol. Evol. 6 , 925–929 (2015).
Artigo Google Scholar
10
Gompert, G. & Buerkle, CA INTROGRESS: um pacote de software para mapear componentes de isolamento em híbridos. Mol Ecol Res 10 , 378–384 (2010).
CAS Artigo Google Scholar
11
Alexander, DH & Lange, K. Aprimoramentos para o algoritmo ADMIXTURE para estimativa de ancestralidade individual. BMC Bioinformatics 12 , 246 (2011).
Artigo Google Scholar
12
Purcell, S. et al . PLINK: um conjunto de ferramentas para associação de genoma inteiro e análises de ligação com base na população. Am J Hum Genet 81 , 559–575 (2007).
CAS Artigo Google Scholar
13
Romano, CM et al . Diversidade viral inter e intra-hospedeiro em um grande surto sazonal de DENV2. PLoS One 8 , e70318 (2013).
PUBLICIDADES CAS Artigo Google Scholar
14
Cugola, FR et al . A cepa brasileira do zika vírus causa defeitos congênitos em modelos experimentais. Nature 534 , 267-271 (2016).
PUBLICIDADES CAS Artigo Google Scholar
15
Costa-da-Silva, AL Cepas de laboratório de Aedes aegypti são competentes para o vírus Zika brasileiro. PLoS One 12 , e0171951 (2017).
Artigo Google Scholar
16
Powell, JR Variação genética em vetores de insetos: morte da tipologia? Insetos 9 , 139 (2018).
Artigo Google Scholar
Baixar referências
Reconhecimentos
O apoio financeiro foi fornecido por doações do Instituto Nacional de Alergias e Doenças Infecciosas dos Estados Unidos, UO1 AI115595, JRP, Pesquisador Principal. BRE era um companheiro de treinamento no NIH 5T32AI007404. JRP recebeu a bolsa Ciência sem Fronteiras do Governo Nacional Brasileiro.
Informação sobre o autor
AfiliaçõesYale University, 21 Sachem Street, New Haven, CT, 06520-8105, EUA
Benjamin R. Evans, Panayiota Kotsakiozi e Jeffrey R. Powell
Departamento de Parasitologia, Instituto de Ciências Biomédicas, Universidade de São Paulo, Av. Prof. Prof. Lineu Prestes, 1374, São Paulo, SP, 05508-000, Brasil
André Luis Costa-da-Silva, Rafaella Sayuri Ioshino, Michele C. Pedrosa e Margareth L. Capurro
Instituto Nacional de Ciência e Tecnologia em Entomologia Molecular, INCT-EM, Rio de Janeiro, Rio de Janeiro, Brasil
André Luis Costa-da-Silva, Rafaella Sayuri Ioshino, Luiza Garziera, Michele C. Pedrosa e Margareth L. Capurro
Moscamed Brasil, Loteamento Centro Industrial São Francisco 9 - lt 15, Juazeiro, BA, 48908-000, Brasil
Michele C. Pedrosa, Aldo Malavasi e Jair F. Virginio
Contribuições
BE e JP desenvolveram tecnologias e realizaram genotipagem; BE e PK realizaram análises e prepararam valores; ALC, RSI e MLC realizaram testes de competência vetorial; LG, MP, AM e JV realizaram os lançamentos e coletaram amostras pós-lançamento; JP concebeu e dirigiu o projeto e redigiu o manuscrito.
autor correspondente
Correspondência para Jeffrey R. Powell .
Declarações de ética
Interesses competitivos
Os autores declaram não haver interesses conflitantes.
Imagem em tamanho grande
Discussão
Nossos dados mostram claramente que a liberação do OX513A levou a uma transferência significativa de seu genoma (introgressão) para a população natural de Jacobina de Ae. aegypti . O grau de introgressão não é trivial. Dependendo da amostra e do critério usado para definir introgressão inequívoca, cerca de 10% a 60% de todos os indivíduos têm algum genoma OX513A (Tabelas 1 e E1).
Uma aparente anomalia nos dados é a aparente diminuição na frequência de indivíduos introgressados entre a amostra de 12 meses e a amostra de 27-30 meses. No entanto, fica claro a partir dos dados de Garziera et al . 6 que a eficácia do programa de liberação começou a diminuir após cerca de 18 meses, ou seja, a população que havia sido bastante reprimida voltou a quase atingir os níveis anteriores à liberação. Especula-se que isso se deve à discriminação de acasalamento contra machos OX513A, um fenômeno conhecido por ocorrer em programas de liberação de machos estéreis 16 . Essa observação também implica que os indivíduos introgrados podem estar em uma desvantagem seletiva, causando sua aparente diminuição após o término da liberação, embora muito mais dados sejam necessários para confirmar isso.
Não se sabe o que impacta a introgressão de uma cepa transgênica de Ae. aegypti possui características importantes para o controle e transmissão de doenças. Testamos OX513A e Jacobina antes da liberação para taxas de infecção por uma cepa de cada um dos vírus da dengue e Zika e não encontramos diferenças significativas (Fig. 3 ). No entanto, isso é para apenas uma cepa de cada vírus em condições de laboratório; sob condições de campo para outros vírus, os efeitos podem ser diferentes. Além disso, a introgressão pode introduzir outros genes relevantes, como para a resistência a inseticidas. A cepa de liberação, OX513A, foi derivada de uma cepa de laboratório originalmente de Cuba, então cruzada com uma população mexicana 7. As três populações que formam a população tri-híbrida agora em Jacobina (Cuba / México / Brasil) são geneticamente muito distintas (dados estendidos Fig. E2 ), muito provavelmente resultando em uma população mais robusta do que a população de pré-liberação devido ao vigor do híbrido.
Esses resultados demonstram a importância de haver um programa de monitoramento genético durante a liberação de organismos transgênicos para detectar consequências inesperadas.
Histórico de mudanças
17 de setembro de 2019
Nota do Editor: os leitores são alertados de que as conclusões deste artigo estão sujeitas a críticas que estão sendo consideradas pelos editores. Uma resposta editorial adicional seguirá a resolução dessas questões.
14 de maio de 2020
Nota do Editor: uma Expressão de Preocupação editorial sobre este artigo foi agora publicada https://www.nature.com/articles/s41598-020-62398-w
Referências
1
Ferguson, NM Desafios e oportunidades no controle de infecções transmitidas por mosquitos. Nature 559 , 490 (2018).
PUBLICIDADES CAS Artigo Google Scholar
2
McGraw, EA & O'Neill, SL Além dos inseticidas: um novo pensamento sobre um problema antigo. Nature Rev Microbiol 11 , 181–193 (2013).
CAS Artigo Google Scholar
3
Thomas, DD, Donnell, CA, Wood, JJ & Alphey, LS Controle de população de insetos usando um sistema genético dominante, repressível e letal. Science 287 , 2474–2476 (2000).
PUBLICIDADES CAS Artigo Google Scholar
4
Phuc, HK et al . Sistemas genéticos letais dominantes de ação tardia e controle de mosquitos. BMC Biol 5 , 11 (2007).
Artigo Google Scholar
5
Carvalho, DO et al . Supressão de uma população de campo de Aedes aegypti no Brasil pela liberação sustentada de mosquitos machos transgênicos. PLoS Negl Trop Dis 9 (7), e0003864 (2015).
Artigo Google Scholar
6
Garziera, L. et al . Efeito da interrupção do alagamento remanescente de mosquitões transgênicos sobre a população silvestre de Aedes aegypi : dois estudos de caso no Brasil. Entomol. Eperimento. Appl. 164 , 327-339 (2017).
Artigo Google Scholar
7
Harris, A. et al . Supressão bem-sucedida de uma população de mosquitos do campo por meio da liberação sustentada de mosquitões machos manipulados. Nature Biotch. 30 , 828–830 (2002).
Artigo Google Scholar
8
Evans, BR et al . Um chip SNP multifuncional de alta capacidade para o mosquito da dengue e da febre amarela. Aedes aegypti. G3 (5), 711–718 (2015).
Google Scholar
9
Frichot, E. & Francois, O. LEA: um pacote R para os estudos da Paisagem e da Associação Ecológica. Methods Ecol. Evol. 6 , 925–929 (2015).
Artigo Google Scholar
10
Gompert, G. & Buerkle, CA INTROGRESS: um pacote de software para mapear componentes de isolamento em híbridos. Mol Ecol Res 10 , 378–384 (2010).
CAS Artigo Google Scholar
11
Alexander, DH & Lange, K. Aprimoramentos para o algoritmo ADMIXTURE para estimativa de ancestralidade individual. BMC Bioinformatics 12 , 246 (2011).
Artigo Google Scholar
12
Purcell, S. et al . PLINK: um conjunto de ferramentas para associação de genoma inteiro e análises de ligação com base na população. Am J Hum Genet 81 , 559–575 (2007).
CAS Artigo Google Scholar
13
Romano, CM et al . Diversidade viral inter e intra-hospedeiro em um grande surto sazonal de DENV2. PLoS One 8 , e70318 (2013).
PUBLICIDADES CAS Artigo Google Scholar
14
Cugola, FR et al . A cepa brasileira do zika vírus causa defeitos congênitos em modelos experimentais. Nature 534 , 267-271 (2016).
PUBLICIDADES CAS Artigo Google Scholar
15
Costa-da-Silva, AL Cepas de laboratório de Aedes aegypti são competentes para o vírus Zika brasileiro. PLoS One 12 , e0171951 (2017).
Artigo Google Scholar
16
Powell, JR Variação genética em vetores de insetos: morte da tipologia? Insetos 9 , 139 (2018).
Artigo Google Scholar
Baixar referências
Reconhecimentos
O apoio financeiro foi fornecido por doações do Instituto Nacional de Alergias e Doenças Infecciosas dos Estados Unidos, UO1 AI115595, JRP, Pesquisador Principal. BRE era um companheiro de treinamento no NIH 5T32AI007404. JRP recebeu a bolsa Ciência sem Fronteiras do Governo Nacional Brasileiro.
Informação sobre o autor
AfiliaçõesYale University, 21 Sachem Street, New Haven, CT, 06520-8105, EUA
Benjamin R. Evans, Panayiota Kotsakiozi e Jeffrey R. Powell
Departamento de Parasitologia, Instituto de Ciências Biomédicas, Universidade de São Paulo, Av. Prof. Prof. Lineu Prestes, 1374, São Paulo, SP, 05508-000, Brasil
André Luis Costa-da-Silva, Rafaella Sayuri Ioshino, Michele C. Pedrosa e Margareth L. Capurro
Instituto Nacional de Ciência e Tecnologia em Entomologia Molecular, INCT-EM, Rio de Janeiro, Rio de Janeiro, Brasil
André Luis Costa-da-Silva, Rafaella Sayuri Ioshino, Luiza Garziera, Michele C. Pedrosa e Margareth L. Capurro
Moscamed Brasil, Loteamento Centro Industrial São Francisco 9 - lt 15, Juazeiro, BA, 48908-000, Brasil
Michele C. Pedrosa, Aldo Malavasi e Jair F. Virginio
Contribuições
BE e JP desenvolveram tecnologias e realizaram genotipagem; BE e PK realizaram análises e prepararam valores; ALC, RSI e MLC realizaram testes de competência vetorial; LG, MP, AM e JV realizaram os lançamentos e coletaram amostras pós-lançamento; JP concebeu e dirigiu o projeto e redigiu o manuscrito.
autor correspondente
Correspondência para Jeffrey R. Powell .
Declarações de ética
Interesses competitivos
Os autores declaram não haver interesses conflitantes.
Informações adicionais
Nota do editor: a Springer Nature permanece neutra em relação a reivindicações jurisdicionais em mapas publicados e afiliações institucionais.
Informação suplementar
Mosquitos transgênicos Aedes aegypti transferem genes para uma população natural
Direitos e permissões
Acesso aberto Este artigo está licenciado sob uma Licença Internacional Creative Commons Atribuição 4.0, que permite o uso, compartilhamento, adaptação, distribuição e reprodução em qualquer meio ou formato, desde que você dê os devidos créditos ao (s) autor (es) original (is) e à fonte, fornecer um link para a licença Creative Commons e indicar se foram feitas alterações. As imagens ou outro material de terceiros neste artigo estão incluídos na licença Creative Commons do artigo, a menos que indicado de outra forma em uma linha de crédito para o material. Se o material não estiver incluído na licença Creative Commons do artigo e seu uso pretendido não for permitido por regulamentação legal ou exceder o uso permitido, você precisará obter permissão diretamente do detentor dos direitos autorais. Para ver uma cópia desta licença, visite http://creativecommons.org/licenses/by/4.0/ .
Reimpressões e permissões
Nenhum comentário:
Postar um comentário